Bitte beachten Sie hierzu ebenfalls die Informationen auf der folgenden Seite: https://wiki.gematik.de/x/v17aI
Labormeldung, LOINC und SNOMED – Fragen und Antworten
Aufbau einer Labormeldung – Meldung eines Erregernachweises nach § 7 Abs. 1 IfSG
Die elektronische Labormeldung basiert auf dem FHIR-Standard, der die Regeln und Spezifikationen für den Austausch von elektronischen Gesundheitsdaten vorgibt.
Wie FHIR und die Terminologiestandards LOINC und SNOMED-CT in DEMIS für die Labormeldung eingesetzt werden, wird auf https://simplifier.net/rki.demis.laboratory und im Implementierungsleitfaden beschrieben.
In Kürze: Die elektronische Labormeldung enthält einen Laborbericht (DiagnosticReport) und ein bis mehrere Erregernachweise (Observations) sowie Angaben zur Probe (Specimen). Der Laborbericht enthält dabei den Meldetatbestand, den Gesamtbefund, auch textlich ergänzbar, sowie die Bewertung „meldepflichtiger Erreger nachgewiesen (PathogenDetected)“ oder „meldepflichtiger Erreger nicht nachgewiesen (PathogenNotDetected)“. Die Erregernachweise beschreiben in codierter Form (LOINC oder SNOMED) den nachgewiesenen Erreger, die verwendete Methode, das Ergebnis, sowie den Befund/die Interpretation. Auch hier können textliche Ergänzungen gemacht werden. Für die zu verwendeten Codes werden Meldetatbestand spezifisch als auch erregerübergreifende Codelisten (ValueSets) zur Verfügung gestellt. Die Probe wird ebenfalls in codierter Form angegeben.
Im Implementierungsleitfaden wird das Lifecyclemanagement beschrieben, also z.B. wie Folgemeldungen und Korrekturmeldungen geschickt werden.
Konkrete Beispiele für Labormeldungen und Informationen für ausgewählte Meldetatbestände sind auf der DEMIS-Wissensdatenbank hinterlegt.
Da die Verwendung der Terminologiestandards LOINC und SNOMED eine entscheidene Rolle in der Meldung spielen, wird dieses Zusammenspiel im Folgenden ausführlich dargestellt.
Regeln der Codierung mit LOINC und SNOMED und Abbildung der Codes in der DEMIS-Schnittstelle
Für die Codierung von Erregern, Material und Methoden der mikrobiologischen Diagnostik werden in DEMIS die internationalen Codier-Standards LOINC und SNOMED verwendet.
LOINC und SNOMED folgen bei der Codierung von Inhalten Regeln, die den Umgang, die Anwendung und Interaktion der Codes beeinflussen. An dieser Stelle sind zentrale Konzepte für das Verständnis von LOINC und SNOMED zusammengefasst sowie Erläuterungen zur Abbildung der Codes in der DEMIS-Schnittstelle gegeben.
1. Codierung von Frage und Antwort
Grundsätzlich kann ein LOINC als die Formulierung einer Frage verstanden werden.
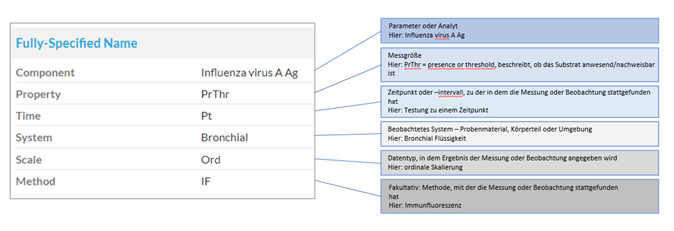
Diese Frage beinhaltet sechs Komponenten (s. Abb 1).
Abb.1 Komponenten zur Beschreibung eines LOINC-Codes nach Regenstrief (s. https://loinc.org/get-started/loinc-term-basics/)
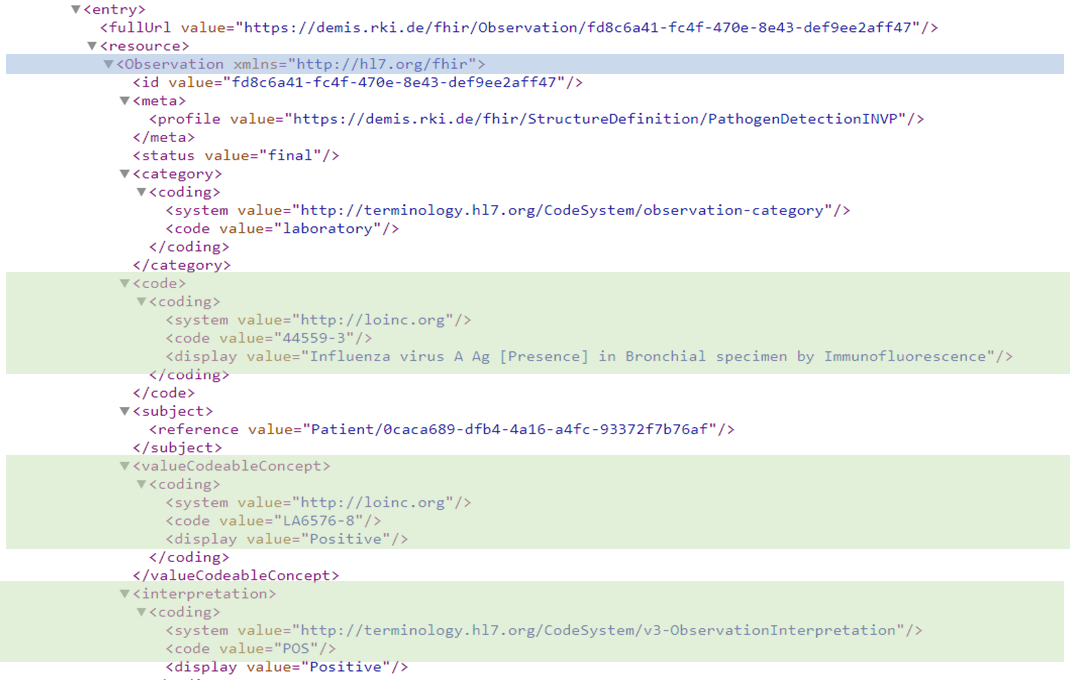
Am Beispiel des LOINC-Codes 44559-3 Influenza virus A Ag [Presence] in Bronchial specimen by Immunofluorescence können die Komponenten wie folgt übersetzt werden:
Abb. 2 Beschreibung der Komponenten des „Fully-Specified Name“ eines LOINC-Codes am Beispiel 44559-3 Influenza virus A Ag [Presence] in Bronchial specimen by Immunofluorescence (s.a. https://www.bfarm.de/DE/Kodiersysteme/Terminologien/LOINC-UCUM/LOINC-und-RELMA/Weitere-Informationen/_node.html)
Ergänzung zur Component (Analytname)
Wenn mit der Messung/Untersuchung mehrere Erreger oder Analyte theoretisch nachgewiesen werden können, dann werden diese aufgezählt und mit einem Pluszeichen ( + ) getrennt, z.B. Mumps virus Ab.IgG+IgM. Das Ergebnis unterscheidet dann nicht zwischen IgG und IgM, es gibt nur ein Gesamtergebnis. Ein weiteres Beispiel ist der Nachweis von Shigatoxingen1 und/oder 2 mit dem Code 80679-4 Escherichia coli Stx1 and Stx2 toxin stx1+stx2 genes [Presence] in Stool by NAA with probe detection. Hier wird nicht zwischen den beiden Genen unterschieden.
Wenn mehrere Analyten getrennt gemessen werden, werden die Analyten durch ein & getrennt, z.B. Salmonella & Shigella & Campylobacter & E. coli sp identified (Long Common name: Salmonella and Shigella and Campylobacter and E. coli sp identified...) Das Ergebnis ist dann entweder Salmonella oder Shigella oder... Solche und vergleichbare Panelcodes sollen in einer DEMIS-Erregernachweismeldungen NICHT verwendet werden, sondern nur erregerspezifische Codes oder die dem Panel untergeordneten Codes für jedes einzelne Ergebnis. Die DEMIS-Meldungen sind Meldetatbestand-spezifisch. Um entsprechend unsaubere Meldungen zu vermeiden, sind gemischte Codes nicht zugelassen.
Jede Frage braucht eine Antwort.
Die Antworten auf eine Frage in Form eines LOINC-Codes können über
- LOINC-Answer-Codes,
- SNOMED-Codes oder
- textuelle Eingaben
gegeben werden.
Für das Zusammenspiel von LOINC-Codes und LOINC-Answer-Codes sowie SNOMED-Codes gelten Regeln. Die „Property“ im LOINC-Code bestimmt, welche Antworten gegeben werden können.
Die Auswahl von LOINC- und SNOMED-Codes ist vielfältig. Mit der nächsten Aktualisierung der Erregernachweismeldung (Version rki.demis.laboratory-1.24.0-rc.1 Vorabveröffentlicht (Stand 01.12.2023)) wird es für alle Antwortmöglichkeiten eigene Answer-ValueSets geben.
Zur Verwendung von LOINC und LOINC-Answer-Codes s.a. „LOINC Users` Guide, Version 2.71, published August 2021“.
2. Abbilden von Frage und Antwort in der Schnittstelle
In der DEMIS-Schnittstelle werden Frage und Antwort wie folgt in der „observation“ abgebildet.
Observation.code Frage in Form eines LOINC-codes
Observation.value Antwort
Es werden drei Möglichkeiten angeboten, unter „Observation.value“ Antworten abzubilden:
- valueCodableConcept - LOINC-Answer-Sets oder SNOMED-Answer-Codes
- valueQuantity + valueQuantityunit - Zahlenwert + Einheit
- valueString - Freitext
Um ein automatisches Auslesen der Meldung und Auswertung (Mapping) aller übermittelten Informationen zu ermöglichen, sollte die Antwort immer codiert als valueCodableConcept oder valueQuantity + valueQuantityunit erfolgen. Freitext-Angaben sind bisher in der Schnittstelle möglich. Mit der nächsten Ausbaustufe der Erregernachweismeldung wird die Verwendung von Freitextangaben unterbunden.
Befund
Ein sehr wichtiger Teil der Meldung ist der Befund. Dieser leitet sich aus der „interpretation“ (Interpretation) in der FHIR-Ressource ab. Sie ist Teil der „observation“ (Erregernachweis). Diese wird nach der HL7 Terminologie codiert: http://terminology.hl7.org/CodeSystem/v3-ObservationInterpretation
Die automatische Übernahme im Gesundheitsamt ist von der korrekten Interpretation/dem Befund abhängig. Typischerweise ist die Interpretation eines Erregernachweises POS (Positive) oder NEG (Negativ), aber insbesondere für Antibiogramme/Resistenzgenbestimmungen muss die Interpretation R (Resistant), S (Susceptible) oder I (Intermediate) sein, es kann auch SDD (Susceptible-dose dependent) verwendet werden.
Beispiel
3. Auslesen der Meldung am Gesundheitsamt
Für das automatische Auslesen der Meldung ist es essentiell, dass der Code für die Frage sowie die möglichen Codes für die Antwort korrekt aufeinander abgestimmt sind und in der Schnittstelle korrekt platziert sind.
Grundlage für die Auswahl von LOINC- und SNOMED-Codes sind die unter https://simplifier.net/rki.demis.laboratory in den ValueSets hinterlegten Codes.
Die ValueSets werden regelmäßig aktualisiert und erweitert und als entsprechende Vorveröffentlichungen veröffentlicht.
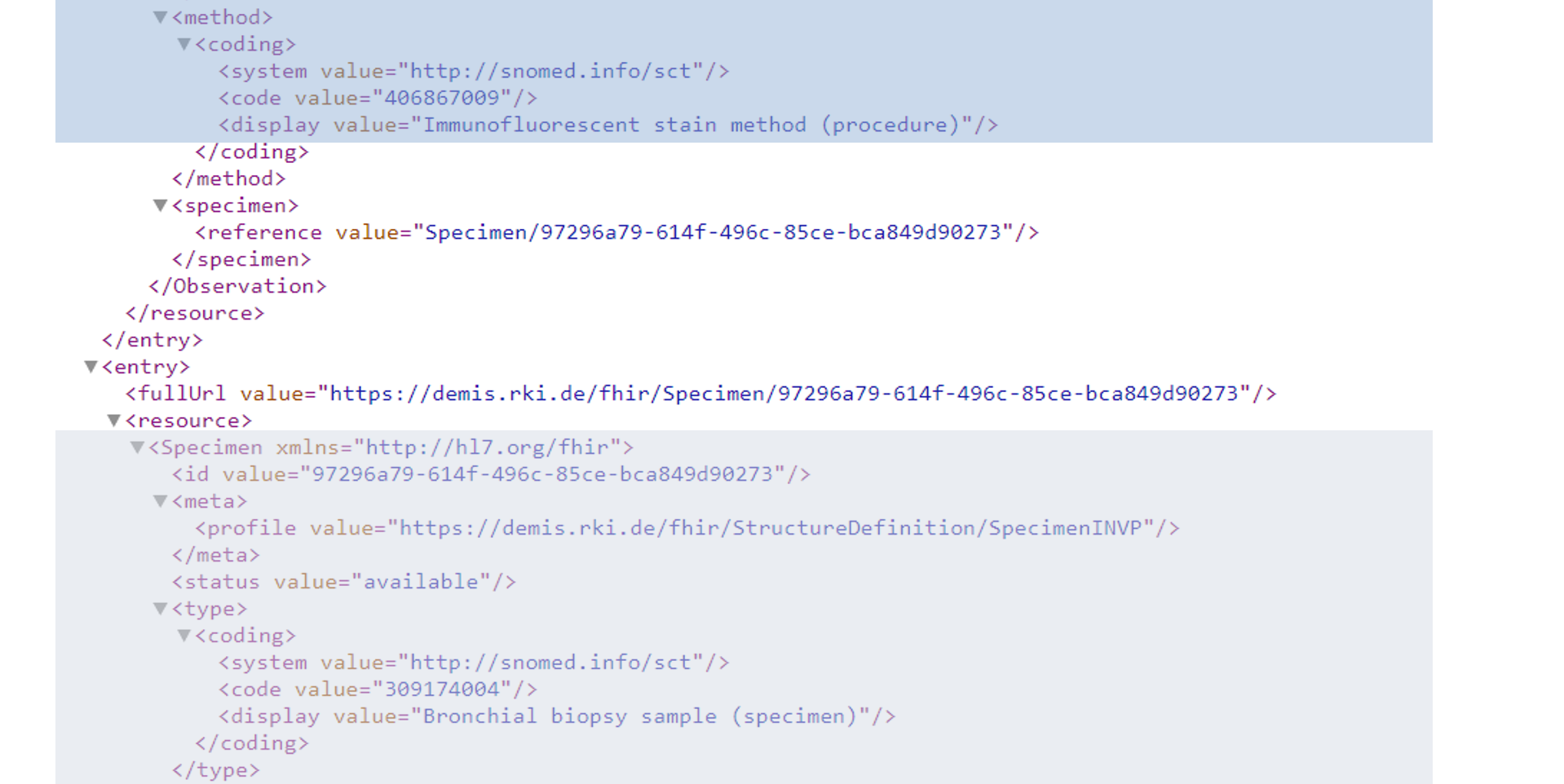
Aus den oben genannten Veröffentlichungen müssen mit der strikten Profilierung ab Januar 2026 auch SNOMED-Codes für Material und Methoden gewählt werden. Der LOINC-Code sollte wenn möglich Material oder Methode nicht spezifizieren. Die Antworten finden Sie hier in der Rubrik "AnswerSet", soweit vorgegeben.
Material und Methode werden in der Schnittstelle unabhängig von der „observation“ abgebildet:
Auch hier muss die Antwort codiert (Specimen.type.code) angegeben werden.
Eine vollständige Auswertung aller Angaben zur von Ihnen durchgeführten mikrobiologischen Diagnostik kann nur erfolgen, wenn die Codierung, das Zusammenspiel der verschiedenen Codes und Abbildung in der Schnittstelle korrekt erfolgen. Wichtig hierbei ist, dass nur die in den ValueSets enthaltenen Codes in den Gesundheitsämtern entsprechend abgebildet und textlich verarbeitet werden können. Wird ein Code verwendet, der nicht in den hinterlegten ValueSets enthalten ist, dann müssen in den Hinweisfeldern (note) entsprechende Ergänzungen gemacht werden, sonst geht aus der Meldung kein Inhalt hervor.